- 所有服務
- 生命科學服務平臺
- 診斷原料開發服務平臺
- 抗體藥物研發服務平臺
同源建模服務系統
背景介紹
生物分子結構同源建模
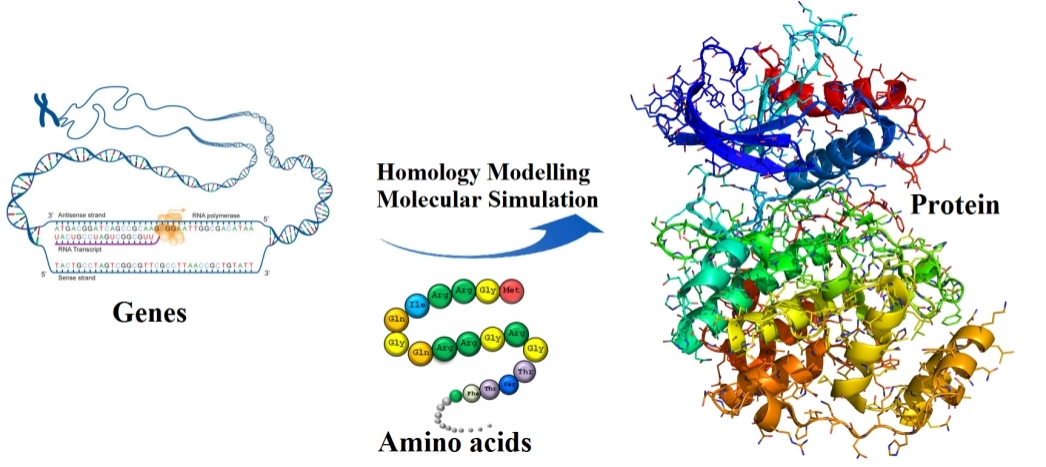
同源模建,蛋白三維結構是理解其生物學功能和進行基于結構的藥物設計的重要基礎。隨著結構生物學的快速發展,使用NMR或X-Ray晶體學等方法測量蛋白三維結構的速度得到了很大提高。然而,由于諸多原因,例如分子量過大或難于結晶,一些蛋白的三維結構仍然難以測定。這種場景下,必須借助計算模擬的手段,來構建蛋白的三維結構。在眾多的計算機建模方法中,同源建模(Homology Modeling)是一種技術最為成熟、準確度最高、結果最為可靠的手段。同源建模(Homology Modeling)的基本假設是:如果未知結構蛋白A與已知結構蛋白B之間具有序列、結構和功能上的同源性,則蛋白A可以以蛋白B為模版來構建其全原子三維結構。
該部分主要包含對蛋白質和小分子結構的構建與優化。對于未知結構的蛋白質分子,在已知基因或氨基酸序列基礎上,通過同源建模方法進行結構預測和同源構建。隨后在建模蛋白質結構基礎之上,利用動力學軟件進行分子動力學模擬,以得到其最優結構模式。
| Modeller Easy Modeller Swift Modeller Discovery Studio Swiss Model I-TASSER HOMCOS … |
圖1 蛋白質同源建模與相關軟件 | |
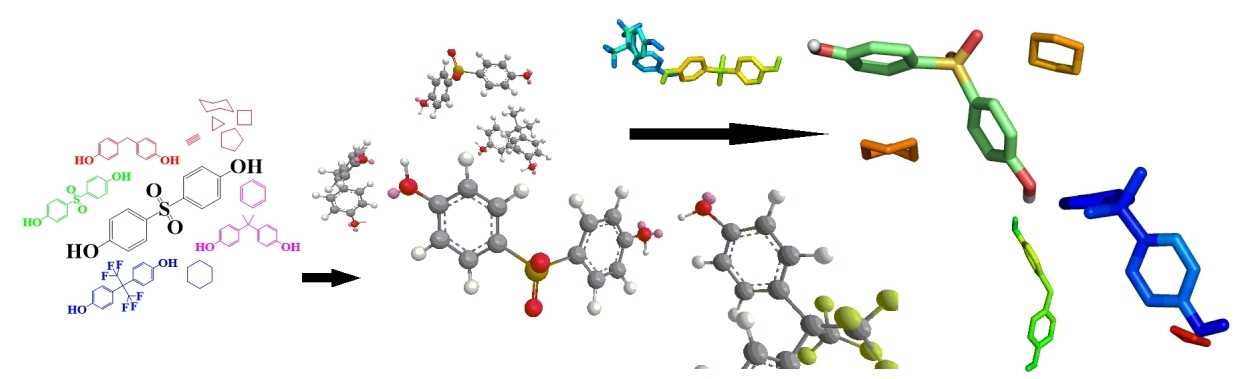
通過經典化學構圖方法對小分子化合物進行結構構建,并采用量化軟件(Gaussian,ORCA等)計算分子電荷分布、分子軌道和反應活化能等對其進行結構優化。
|
圖2 小分子模型構建 |
構建同源模型的流程通常都是簡潔而清晰的。首先,通過序列相似性搜索工具為查詢序列(Query)搜索序列相似程度較高的已知結構蛋白作為候選模版。其次,將查詢序列與候選模版進行比對對齊(Alignment),以序列相同度(Indentity)為指標,從中挑選一個或多個模版組合作為最終模版。自此進入建模的實際階段:比對對齊后,那些與模版序列完全相同的殘基所有原子座標完全拷貝自模版,而那些相似殘基僅有骨架原子取自模版——側鏈座標另行構建,那些缺失序列則通過查找一個Loop 數據庫來進行補缺;最后,這個虛擬的預測模型在特定的勢場參數下進行能量優化,得到最終模型。
研錦生物提供高質量的同源建模服務,其結果可以幫助研究人員了解目標蛋白的結構性質,分析其“結構—功能”關系,并可用于藥物篩選、發現某種疾病的潛在治療藥物,或者研究靶標與藥物的相互作用模式等。